
自单细胞测序技术出现以后,各种单细胞的分析软件及方法也应运而生。由于技术的高速发展,并且在个性化药物治疗设计等方面开始越来越多的应用,现阶段出现了很多单细胞的测序平台和分析方法。而面对各种各样的算法,如何选择合适的分析方法对数据进行分析使其发挥最大价值仍是当前面临的一个重大挑战。
近日,美国洛马林达大学基因组学中心的王昌宏教授(Charles Wang)和美国食品药品管理局(FDA)的肖文明博士共同作为通讯作者在Nature Biotechnology发表了题为“A multicenter study benchmarking single-cell RNA sequencing technologies using reference samples”的研究性文章,该项研究重点研究了不同生物信息处理方法对数据的影响,评估了不同中心的不同测序平台上的单细胞测序性能。这是一项综合性研究,涉及不同技术平台、不同样品和生物信息学方法(包括预处理、归一化和批次效应校正),为平台优化和软件的选择提供了实践指导,也为分析流程的开发提供了重要资源。

该研究使用了四种scRNA-seq平台:包括10x Genomics,Fluidigm C1, Fluidigm C1 HT和Takara Bio ICELL8;测序工作分别在四个研究中心进行:洛马林达(LLU),美国国立癌症研究所(NCI), 美国食品药品监督管理局(FDA)和美国Takara Bio(TBU)。对于样本的选择,研究人员选择了两个特征明显的参考细胞系:来自同一供者的乳腺癌细胞系(样本A,HCC1395)和正常B淋巴细胞系(样本B, HCC1395BL)。然后使用3 '或全长单细胞转录组测序方法对单细胞进行了测序,建立了20套标准单细胞转录组数据。
对于生成的20个数据集,研究人员对不同的数据预处理方法、数据归一化方法、批次效应校正方法等进行了评估。研究人员比较了六种单细胞数据预处理方法(10×: Cell Ranger, UMI-tools, zUMIs; C1&ICELL8: featureCounts, RSEM, kallisto),评估了八种不同的归一化方法(sctransform, scran deconvolution, counts per million (CPM), logCPM, trimmed mean of M values (TMM), DESeq, quantile, Linnorm)和七种不同的批次校正算法(Seurat3, fastMNN, Scanorama, batch-balanced k-nearest neighbors (BBKNN), Harmony, limma, ComBat )。总体研究设计如下图:
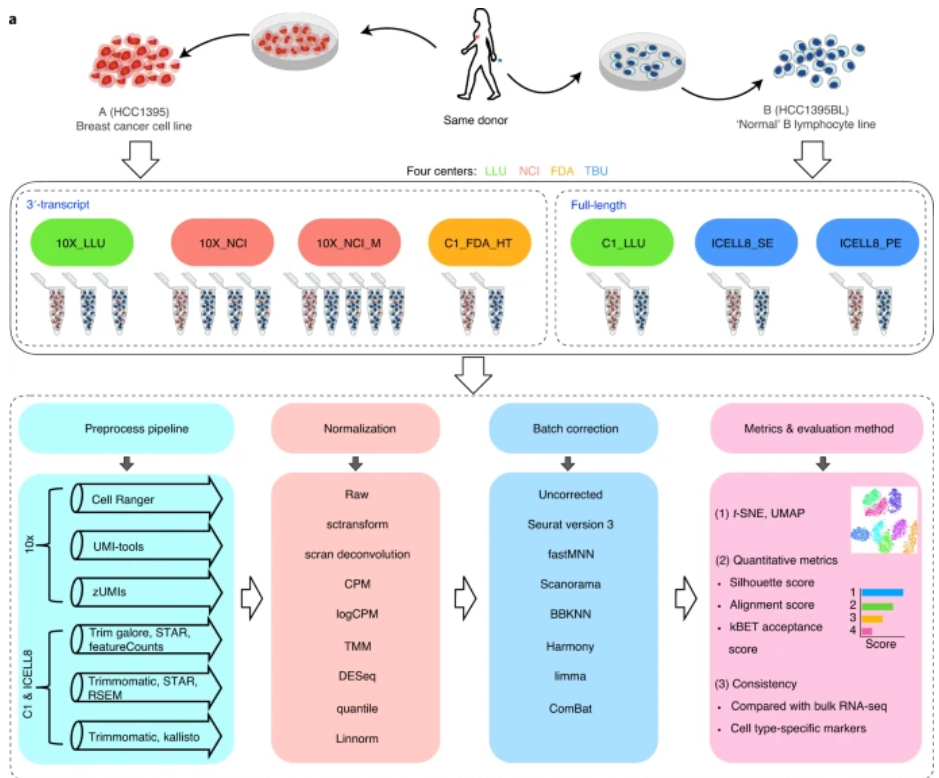
主要结果
1
●
数据预处理方法的比较
对基于UMI(Unique Molecular Identifier)的scRNA-seq数据,研究人员比较了三种预处理方法:Cell Ranger(10x Genomics)、UMI-tools和zUMIs。结果显示,三种方法在鉴定细胞数量和每个细胞检测到的基因数量层面均存在差异。对非基于UMI的scRNA-seq数据,他们比较了另外三种预处理方法:featureCounts、RSEM和kallisto。这些数据预处理流程包括修剪、比对和基因计数。结果表明,三个不同的预处理方法检测到的基因数量的差异比较大。kallisto在全长转录本scRNA-seq数据集中发现了每个细胞中更多的基因。
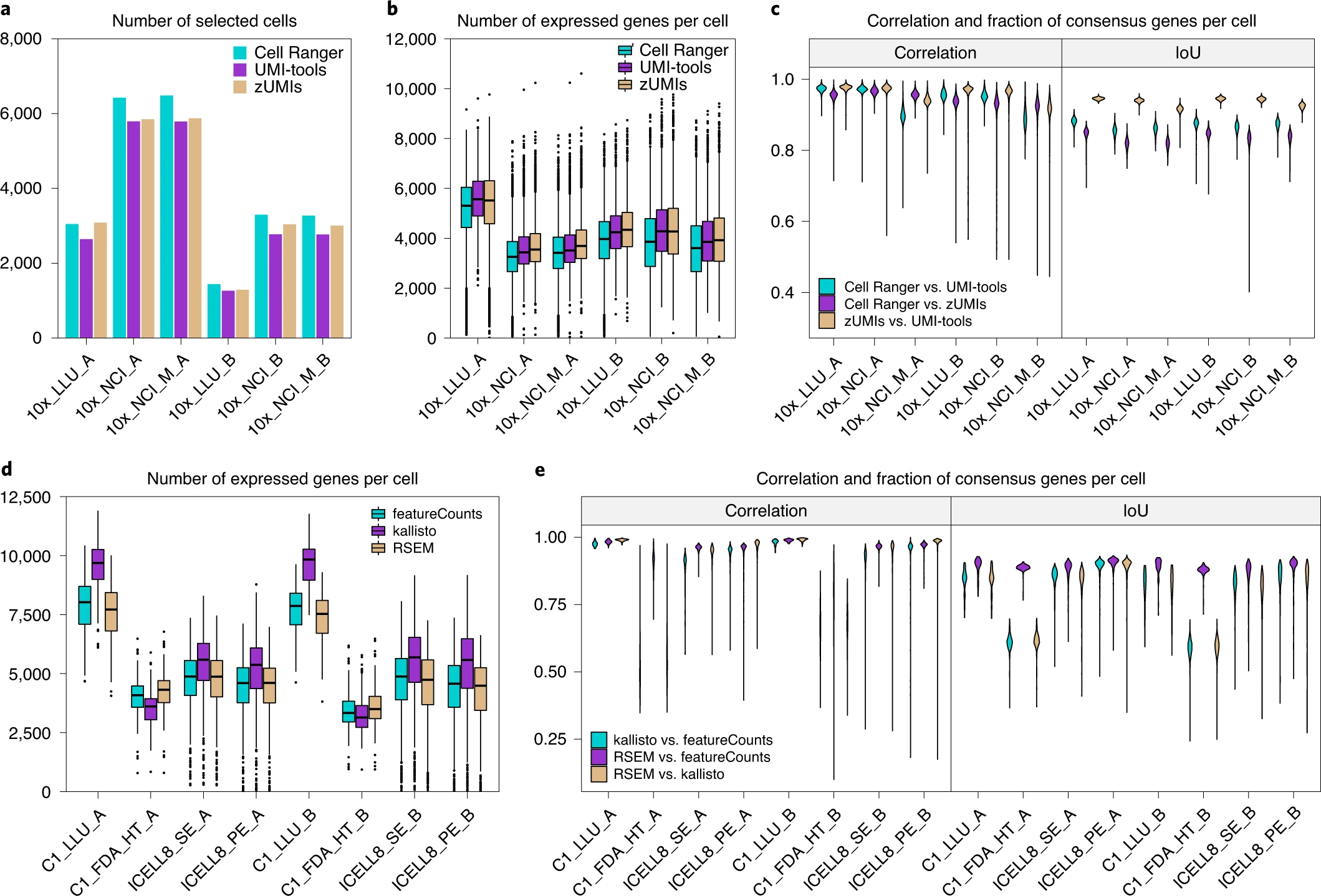
2
●
归一化方法的比较
研究人员使用Sihouette指标评估归一化算法的效果和聚类结果。TMM和quantile未能使样本A和样本B归一化,其Sihouette得分与对照的原始数据相似,TMM和quantitle结果最差,因此不建议使用;而Sctreansform 流程处理后的数据方差最小。
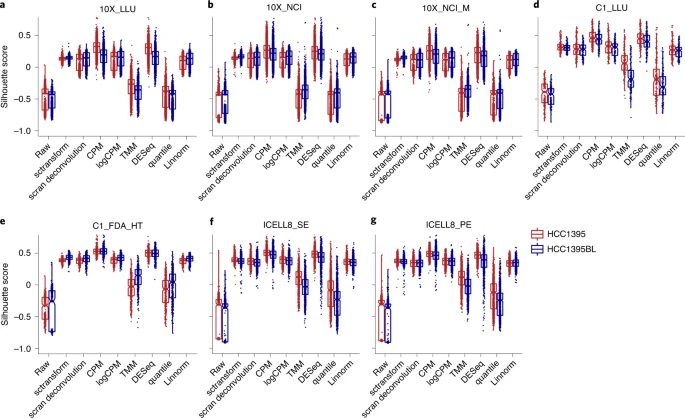
3
●
不同批次效应校正方法的比较
研究人员通过四种不同的数据集组合来评估这些算法的性能,方案1包含所有单细胞转录组数据集,包括混合和纯合数据集,用以评估算法的clusterability(分离不同细胞类型的能力,即不同的细胞类型是否能够很好的区分开);方案2包含了乳腺癌细胞系数据(样品A),用以评估算法的mixability(对相似细胞类型进行分组的能力,即不同批次的相同细胞类型是否能够聚类到一块);方案3包含B细胞系来源数据,评估软件的mixability;方案4中,将5%或10%的乳腺癌细胞(样本A)加入到B淋巴细胞(样本B)中,用10x Genomics平台横跨两个中心测序得到,用以评估算法的clusterability。
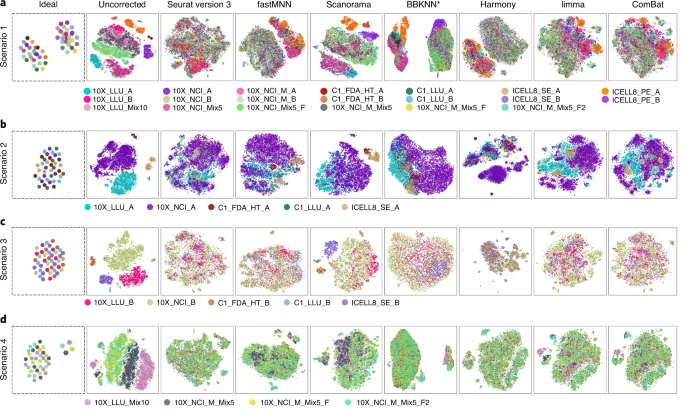
方案1的结果显示,在消除批次效应和两种细胞类型分离方面,BBKNN(在聚集性方面排名最高)、fastMNN和Harmony最有效;方案2和方案3的结果显示,Seurat3是将不同批次的相似细胞聚集在一起的最佳方法之一。而当仅分析来自10x平台的数据时,Scanorama都能清楚地分离了不同的细胞,并且将相似的细胞分组在一起。
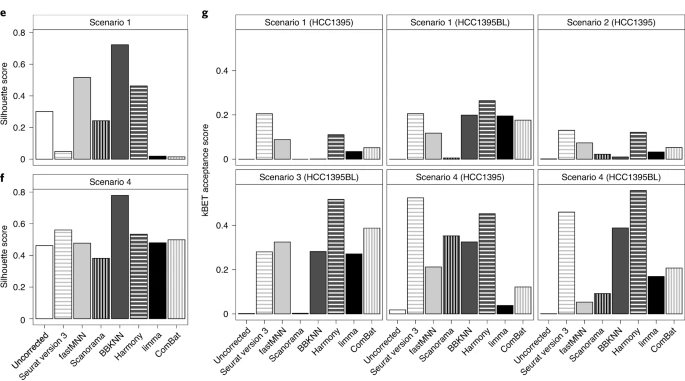
研究表明,尽管数据预处理对单细胞基因检测和细胞分类有一定的影响,但批次效应的处理才是迄今为止正确分类细胞的最关键步骤。另外,通过混合两种细胞系,该研究还评估了样本及数据的组成和性质对生物信息学方法分析结果的影响。
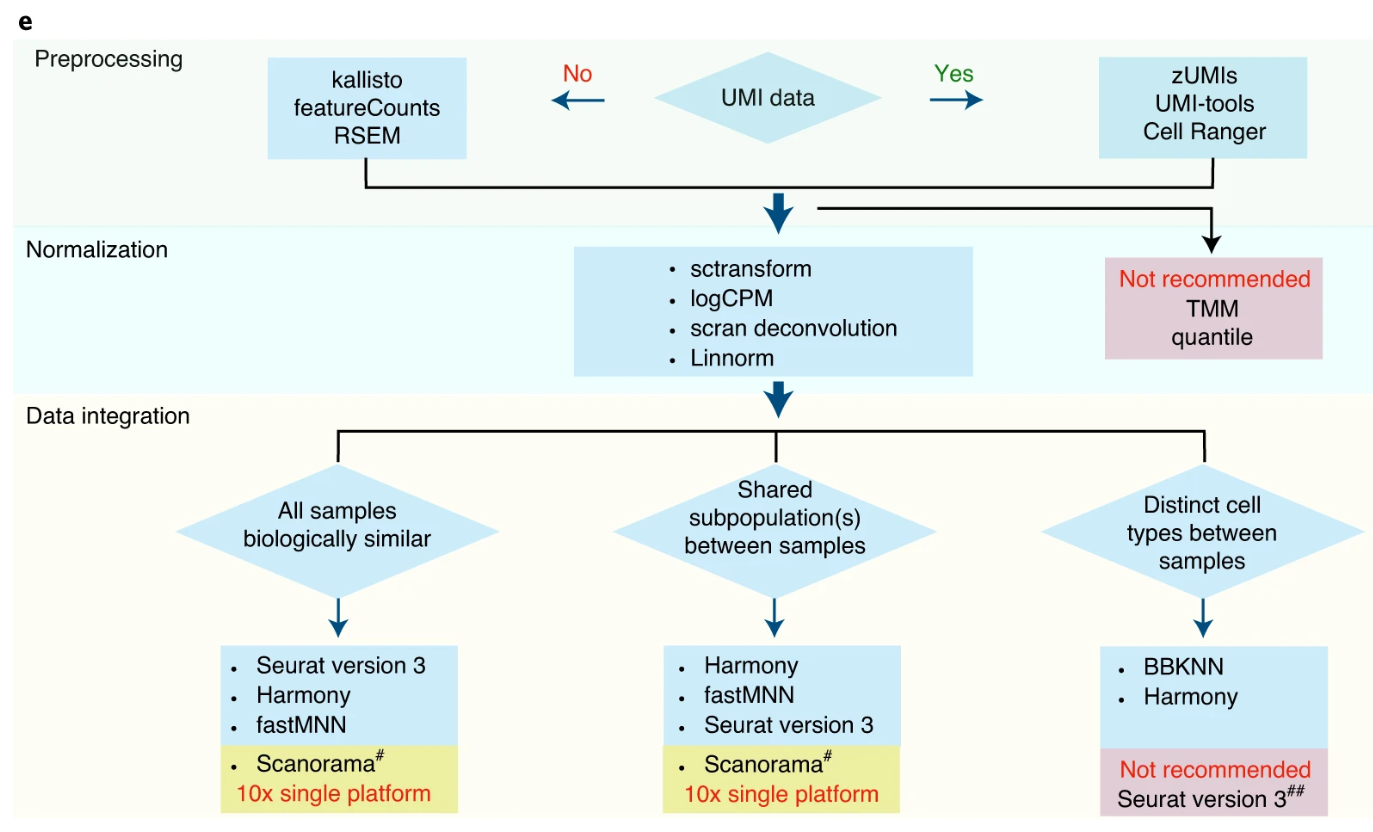
研究人员对这些预处理方法和算法进行了综合排序,如下图所示,基于10×的数据可以用文中所列的任何方法进行预处理,而kallisto则更适用于全长转录组测序数据的预处理。

下图为本研究中提出的scRNA-seq分析的最佳实践建议。仅分析10×数据时,Scanorama的效果就很好。Seurat版本3适用于生物学上相似的样品,但如果在不同批次中存在很大比例的不同细胞类型,则批次效应过度校正,且细胞类型分类错误。
综上,该研究比较分析了6种预处理流程、8种归一化方法和7种批次校正算法,结果表明,单细胞转录组数据之间确实有批次效应。同时,该研究强调了测序技术平台和分析数据算法的重要性,也为科研人员选择最适合解决科学问题的技术平台和生物信息方法提供了实践指导。
参考文献:
Chen, W., Zhao, Y., Chen, X. et al. A multicenter study benchmarking single-cell RNA sequencing technologies using reference samples. Nat Biotechnol (2020).
原文链接:https://www.nature.com/articles/s41587-020-00748-9
 微信公众号
微信公众号
