
01
细胞内RNA-RNA相互作用的重要功能
人类基因组通过广泛转录产生大量的编码蛋白质的RNA和缺乏蛋白质编码潜力的非编码RNA (ncRNA) 。在快速增殖的哺乳动物细胞中,按质量计算总转录本中超过95%的均为ncRNA,如rRNA、tRNA、microRNA和长链非编码RNA (lncRNA)。相比之下,剩下不到5%的才是编码蛋白质的mRNA [1]。与mRNA相比,ncRNA主要通过形成复杂的三级结构,然后通过互补碱基配对或非互补的近端接触与其他mRNA或ncRNA相互作用来实现其生物学功能。
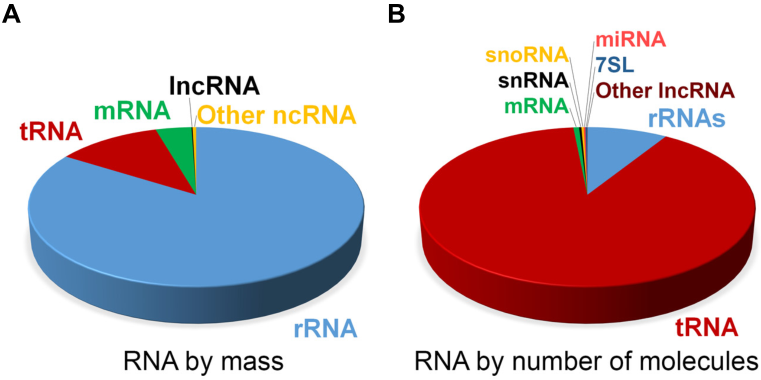
哺乳动物细胞中RNA丰度的评估 [1]
研究表明,近端RNA-RNA相互作用(分子内和分子间)在许多重要的生物过程中发挥着重要的调节作用,如转录、pre-mRNA剪接和蛋白质翻译。例如,小核RNA (snRNA)和剪接信号之间的分子间RNA-RNA相互作用是盒式外显子包含或跳过的主要决定因素,RNA的结构与它们的加工和功能密切相关。剪切复合体的核心组分之一U1 snRNA可以通过与新生出来的pre-mRNA发生直接相互作用从而调控转录进程[2]。

U1 snRNA与pre-mRNA直接相互作用调控转录进程 [2]
靶向β-分泌酶1(BACE1)的lncRNA在阿尔茨海默病的病理学中发挥作用,反义lncRNA BACE1-AS通过与其mRNA互作稳定它的结构来调节有义BACE1基因(BACE1-S)的表达。BACE1-AS在阿尔茨海默病中升高,增加BACE1蛋白的数量,随后增加了β-淀粉样肽的产生[3]。
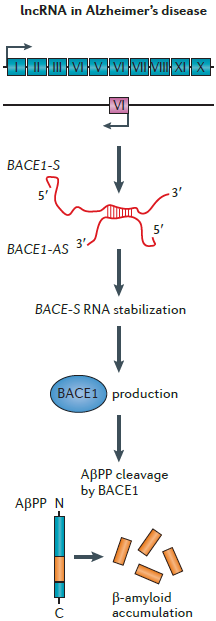
反义lncRNA BACE1-AS与mRNA BACE1-S互作调节BACE1-S的表达 [3]
在Hela细胞中,原癌基因MYC的启动子RNA可以与远端增强子转录的eRNAs相互作用,调控染色质loop形成,进而介导hnRNPK蛋白及其二聚体将Pol II从超级增强子CCAT1-5L位点递送到MYC的启动子从而激活MYC的转录[4]。
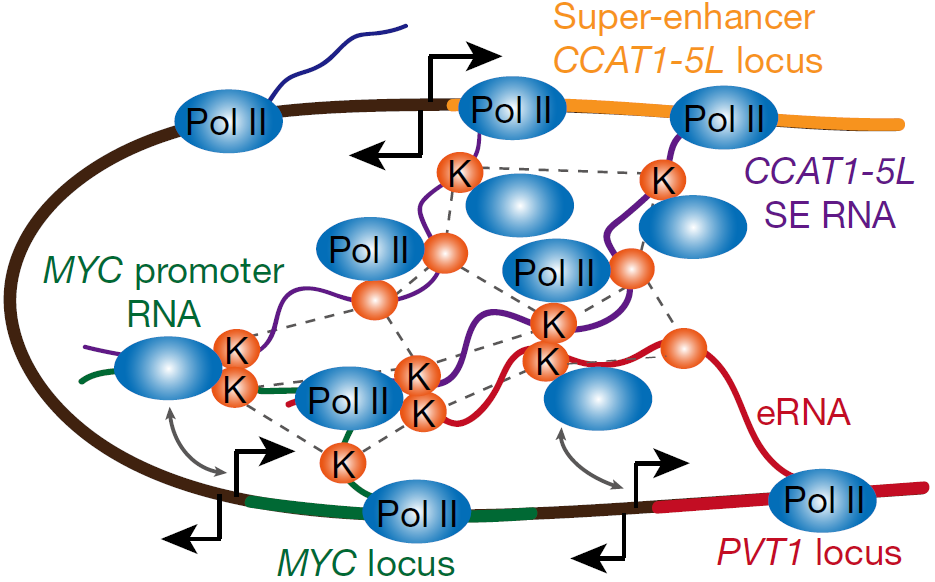
CCAT1-5L与MYC启动子RNA和eRNAs相互作用介导染色质环形成激活MYC转录的调控模型 [4]
分子内RNA-RNA相互作用,特别是duplexes(两条互补链形成的双链RNA)是形成复杂高级结构的基石,这些相互作用已经被证明在很大程度上是通过自身互补、和/或各种RNA结合蛋白(RPB)介导和维持的。RPB广泛参与RNA结构形成,最近的RNA pull down和质谱分析发现超过1500个RPB可以结合、引导和调控活细胞中的各种RNA,关于RPB如何组织RNA结构组(生物体转录组的结构信息)和相互作用组,是近年来科学家们的研究重点。为了识别RBP所结合的RNA序列,RIP (RNA免疫沉淀)和CLIP(交联和免疫沉淀)这两种方法都使用特异性抗体拉下目标RBP-RNA复合物并对结合的RNA进行测序。然而,这两种方法只检测特定RBP与不同RNA结合的情况,并没有分析这些RBP是否介导了潜在的分子内或分子间RNA-RNA相互作用。
02
检测细胞内RNA-RNA相互作用的技术方法
为了鉴定RNA-RNA相互作用及RNA双链结构,在过去的几十年里,各种计算模型、进化共偶联分析和化学或基于酶的探测方法得到了迅速发展,很多高通量的研究细胞全范围内RNA-RNA相互作用的方法也被人们开发出来,包括但不限于CLASH、hiCLIP、RPL、SPLASH、MARIO、RIC-seq等技术。这些方法极大地促进了人们对RNA结构在各种生物学过程如RNA的折叠、RNA-蛋白相互作用和RNA代谢中的理解。
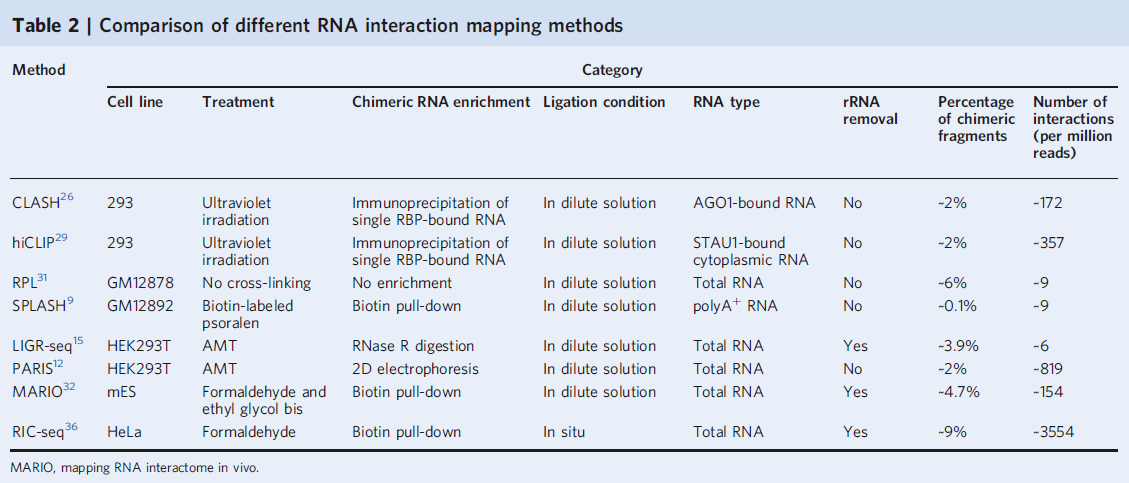
不同RNA互作技术的比较 [5]
此外,RNA互作技术还可以用于绘制RNA病毒基因组的空间构象,近日有研究者使用Simplified SPLASH方法绘制了SARS-CoV-2在生命周期的体内RNA-RNA相互作用,提供了转录调节序列(TRS-L 和 TRS-Bs)之间相互作用的第一个直接证据,发现其基因组 RNA 经历了强烈的压缩,但基因组结构域保持稳定,结构域特征信号增强,全局环化发生减弱[6]。还有研究者采用vRIC-seq技术重构了SARS-CoV-2 RNA基因组的三维结构模型[7]。这些研究成果为病毒的复制、不连续转录和翻译移码的调节提供了结构基础,有助于人们开发出更好的抗病毒策略。
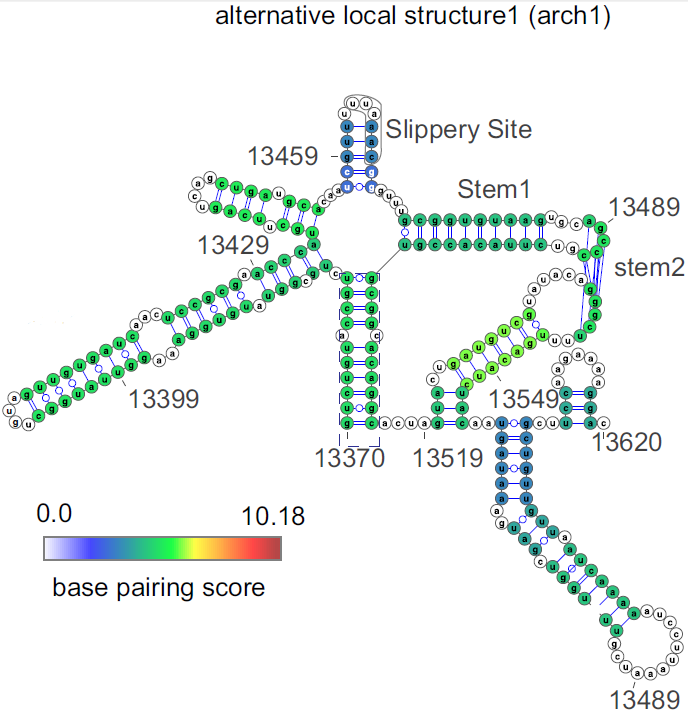
SARS-CoV-2 RNA的局部可变结构 [6]
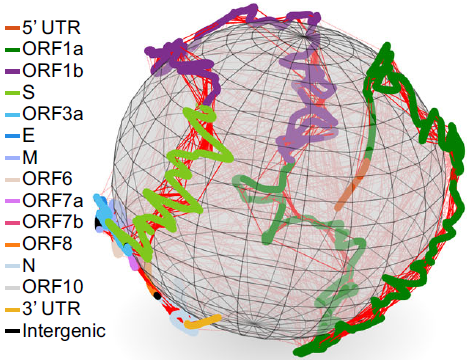
SARS-CoV-2 RNA的全局三维结构模型 [7]
然而,这些方法在广泛应用层面也存在一些局限性,例如CLASH、hiCLIP、PARIS、LIGR-seq所获得的嵌合序列的覆盖率都较低(<4%),且邻近连接步骤是在稀释溶液中进行的,可能会导致一些假阳性。RPL技术由于缺乏交联和嵌合富集,仅限于获得分子内的RNA相互作用。MARIO通过半胱氨酸残基中的巯基基团将蛋白质生物素化,然后将蛋白质-RNA复合物固定在磁珠上进行近距离连接,在细胞裂解液中生物素化的效率会降低,可能会导致一些蛋白质-RNA复合物的丢失。

MARIO (Mapping RNA interactome in vivo) [8]
RIC-seq通过微球菌核酸酶消化部分裸露的RNA,使用pCp -生物素标记RNA,将空间邻近的RNA原位连接起来形成嵌合RNA分子,减少了稀释体系中进行连接带来的可能假阳性,提高了嵌合RNA序列占双端唯一比对的比例(~9%,rRNA-)及(~16%,rRNA+)。然而此实验过程中过于频繁的离心清洗步骤可能会造成一些RNA-RNA互作的丢失,整个实验耗时相对较长(~6天)。

RIC-seq (RNA in situ conformation sequencing) [5]
03
永利集团3044Hi-R技术检测细胞内RNA-RNA相互作用
基于这些技术的特点,永利集团3044综合吸取各自的优势,凭借已有的Hi-C实验技术基础,通过调整和简化实验流程,开发出可用于获得RNA-RNA空间互作的新技术,命名为Hi-R(High-throughput RNA Conformation Capture)技术,并使用Hela细胞进行了两轮测试,均获得了高达15%的嵌合RNA序列(rRNA+),成功实现了获得RNA-RNA空间互作信息的目标。
|
永利集团3044Hi-R测试数据(rRNA+) |
Hela-HiR-Lib1 |
Hela-HiR-Lib2 |
|
Number of input reads |
6736493 |
8628015 |
|
Average input read length |
150 |
150 |
|
UNIQUE READS: |
||
|
Uniquely mapped reads number |
4190705 |
4718211 |
|
Uniquely mapped reads % |
62.21% |
54.68% |
|
Average mapped length |
139.01 |
122.68 |
|
Number of splices: Total |
150282 |
35448 |
|
Number of splices: Annotated (sjdb) |
89264 |
6628 |
|
Number of splices: GT/AG |
90610 |
7028 |
|
Number of splices: GC/AG |
876 |
151 |
|
Number of splices: AT/AC |
233 |
60 |
|
Number of splices: Non-canonical |
58563 |
28209 |
|
Mismatch rate per base, % |
2.33% |
3.28% |
|
MULTI-MAPPING READS: |
||
|
Number of reads mapped to multiple loci |
323412 |
206491 |
|
% of reads mapped to multiple loci |
4.80% |
2.39% |
|
Number of reads mapped to too many loci |
3434 |
1666 |
|
% of reads mapped to too many loci |
0.05% |
0.02% |
|
UNMAPPED READS: |
||
|
Number of reads unmapped: too short |
1763408 |
3395099 |
|
% of reads unmapped: too short |
26.18% |
39.35% |
|
Number of reads unmapped: other |
2724 |
1275 |
|
% of reads unmapped: other |
0.04% |
0.01% |
|
CHIMERIC READS: |
||
|
Number of chimeric reads |
647337 |
741044 |
|
% of chimeric reads / Uniquely mapped reads |
15.45% |
15.71% |
|
% of chimeric reads / Number of input reads |
9.61% |
8.59% |
至此,永利集团3044不仅可以提供DNA水平的DNA-DNA空间互作信息Hi-C技术,还可以提供RNA水平的RNA-RNA空间互作信息Hi-R技术,表观类的产品类型得到进一步丰富,现已包括:
Hi-C、Promoter Capture Hi-C、Meta 3C/Meta Hi-C、低起始量Hi-C、Hi-R、ATAC-seq、低起始量ATAC-seq、组蛋白CUT&Tag、转录因子/非组蛋白CUT&Tag、WGBS、Tn5-FISH等多组学技术研究方案,欢迎各位老师咨询与合作~
配图来源于网络/侵删
参考文献:
[1] Palazzo AF and Lee ES. Non-coding RNA: what is functional and what is junk? 2015, Front. Genet.6:2. doi: 10.3389/fgene.2015.00002.
[2] Engreitz, et al. RNA-RNA interactions enable specific targeting of noncoding RNAs to nascent Pre-mRNAs and chromatin sites. 2014, Cell. 159(1):188-199. doi: 10.1016/j.cell.2014.08.018.
[3] Esteller M. Non-coding RNAs in human disease. 2011, Nature Reviews | Genetics. 12(12):861-74. doi: 10.1038/nrg3074.
[4] Cai et al. RIC-seq for global in situ profiling of RNA-RNA spatial interactions. 2020, Nature. 582(7812):432-437. doi: 10.1038/s41586-020-2249-1.
[5] Cao et al. Global in situ profiling of RNA-RNA spatial interactions with RIC-seq. 2021, Nature Protocol. 16(6):2916-2946. doi: 10.1038/s41596-021-00524-2.
[6] Zhang et al. In vivo structure and dynamics of the SARS-CoV-2 RNA genome. 2021, Nature Communications. https://doi.org/10.1038/s41467-021-25999-1.
[7] Cao et al. The architecture of the SARS-CoV-2 RNA genome inside virion. 2021, Nature Communications. 12(1):3917. doi: 10.1038/s41467-021-22785-x.
[8] Nguyen et al. Mapping RNA–RNA interactome and RNA structure in vivo by MARIO. 2016, Nature Communications. 7:12023. doi: 10.1038/ncomms12023.
 微信公众号
微信公众号
