
TIPS
全文共5000字,阅读需要15分钟,建议先收藏后细看。转发此文至朋友圈并截图发至公众号后台,可领取已发表T2T基因组文献。
转眼已是年末,当我们回顾2021年基因组学研究的成果与荣誉时,不难发现“Gap Free、T2T、完成图”已成为我们绕不开的话题,而这也是诸多科研学者在回顾和总结动植物基因组20周年时最看好的方向。此外,2021年科研学者也先后发表了第一个高等动物(人)、第一个高等植物(水稻)的基因组完成图,我们可以将2021年定义为“Gap free基因组元年”。基于此,小编对2021年基因组完成图的研究进展进行初步总结,以期能使更多科研学者领略基因组完成图的“激情与魅力”。
表1 已发表基因组完成图/近完成图汇总

基因组完成图-植物篇
自2001年Nature刊登拟南芥基因组以来,植物基因组测序已经走过了整整20年,这期间已有超900+物种、1200+文章发表(数据截止2021年12月),但关于植物基因组完成图的报道却寥寥无几。目前已构建基因组完成图/近完成图的植物包括水稻、拟南芥、香蕉、澳洲胡桃、玉米、大麦等,具体研究概况如下。
01 水稻基因组完成图

(一)
文章题目:Two Gap-free Reference Genomes and a Global View of the Centromere Architecture in Rice
发表期刊:Molecular Plant
完成图构建策略:PacBio HiFi+CLR
研究结果:研究者采用高深度的HiFi和CLR测序,组装出0 gap的ZS97和MH63 R3版本参考基因组(基因组大小分别为391.56Mb和395.77Mb)。基于Gap free的参考基因组,研究者对水稻12条染色体上着丝粒区域的结构和功能进行了详细研究,发现着丝粒核心区域的长度在不同染色体上存在10倍差距。在ZS97和MH63着丝粒区域,研究者分别鉴定到了395个和539个非TE基因,但其转录活性低、特异性表达的比例低,且大多数活跃转录的基因位于着丝粒周围区域。此外,研究者还发现相同染色体中CentO重复序列的相似性高于跨染色体的相似性;亚洲水稻的同一亚种(或自然群体)中,同一染色体着丝粒核心区域中的CentO卫星重复序列的长度在不同品种之间有显著差异。
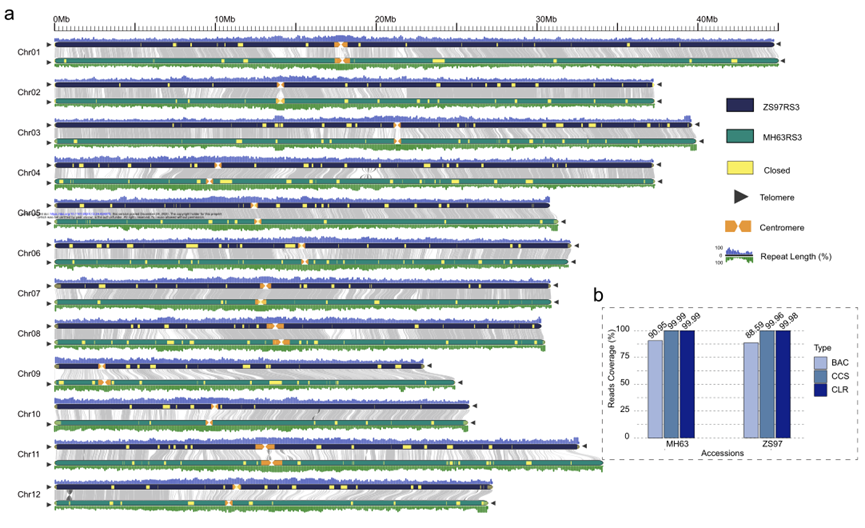
图1 Gap free的ZS97和MH63参考基因组
(二)
文章题目:Gapless indica rice genome reveals synergistic effects of active transposable elements and segmental duplications that promote rice genome evolution
发表期刊:Molecular Plant
完成图构建策略:PacBio HiFi
研究结果:基于HiFi组装、与参考基因组比较、补gap等方式,研究者构建了明恢63(MH63)的基因组完成图MH63KL1,其包括12条Contig(Contig N50=31.93Mb),分别对应水稻的12条染色体。通过与粳稻相比,研究者发现MH63KL1(籼稻)具有更多的节段重复(SD)和转座子(TE)序列,并证实了重复序列可以产生新基因并促使新基因进行适应性进化。此外,节段重复可以产生大量重复基因并以剂量效应的方式影响水稻的相关性状,而转座子的插入既可以影响重复基因的表达,还可以增加重复基因的进化。总之,节段重复和转座子可以协同来促进水稻基因组的进化。
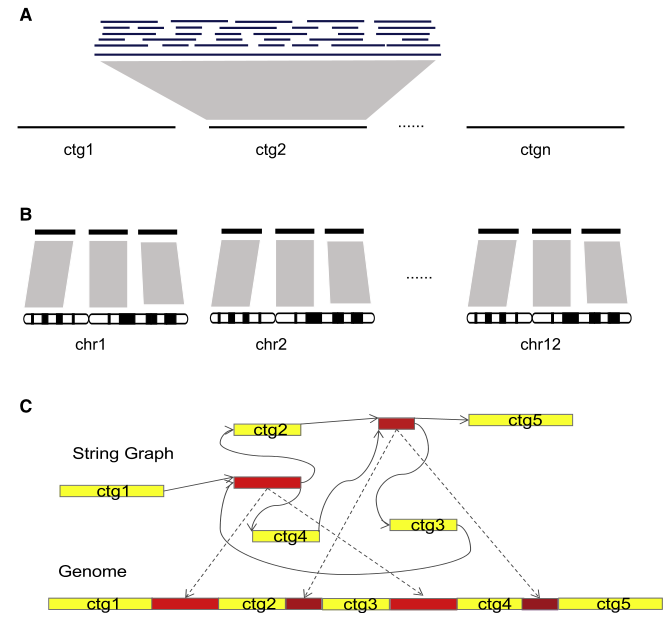
图2 水稻MH63无缺口基因组的组装策略
02 拟南芥基因组完成图

(一)
文章题目:The genetic and epigenetic landscape of the Arabidopsis centromeres
发表期刊:Science
完成图构建策略:PacBio HiFi+ONT Ultra-Long+Bionano
研究结果:通过PacBio HiFi和ONT超长测序,研究者构建了包含5个着丝粒的拟南芥近完成图。通过鉴定着丝粒上的CEN180序列(包含180个碱基的卫星重复序列),研究者发现不同染色体上CEN180序列存在显著差异,而同一条染色体内的CEN180序列呈现均质化趋势。拟南芥5号染色体着丝粒的CEN180序列含量很低(是其它着丝粒的12%-22%),这主要是逆转录转座子ATHILA入侵促进了其序列的变异,进而影响了拟南芥5号染色体着丝粒内CEN180序列的均质化过程。此外,与近着丝粒区域相比,拟南芥着丝粒区域增加了常染色质修饰,降低了异染色质修饰,这说明近着丝粒区域和着丝粒区域共同形成一个混合的染色质状态。总之,拟南芥CEN180序列的均质化和多样化共同推动了其着丝粒结构和功能的进化。
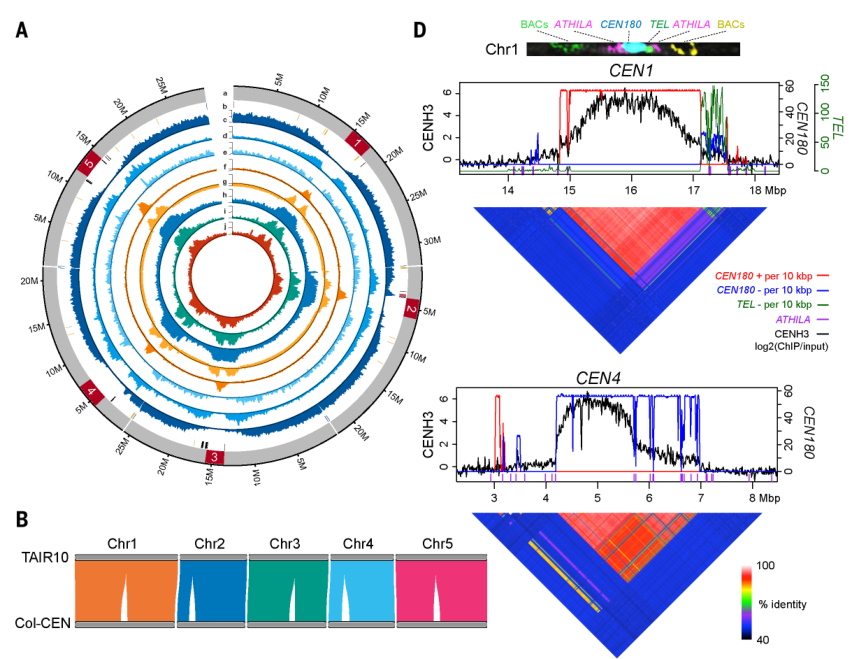
图3 拟南芥着丝粒的近完成图
(二)
文章题目:High-quality Arabidopsis thaliana Genome Assembly with Nanopore and HiFi Long Reads
发表期刊:Genomics,Proteomics & Bioinformatics
完成图构建策略:PacBio HiFi+ ONT Ultra-Long+Hi-C
研究结果:基于PacBio HiFi、ONT ultra-long和Hi-C技术,研究者报道了仅包含两个gap的拟南芥近完成图(Col-XJTU),与TAIR10.1参考基因组相比,引入了14.6 Mb的新序列,且XJTU装配的5条染色体具有更高的准确性(QV>60)。5条染色体中,3号和5号染色体为T2T,4号染色体除了核仁区域也全部组装完成,而1号和2号染色体由于着丝粒区域富含CEN180序列,研究者分别组装出3.5M和4M的着丝粒序列。通过着丝粒结构和表观遗传学分析,研究者发现CENH3对CEN180表现出强烈的偏好,且在CENH3富集区域观察到低甲基化模式。总之,Col-XJTU为拟南芥着丝粒结构和功能的研究提供了新见解。
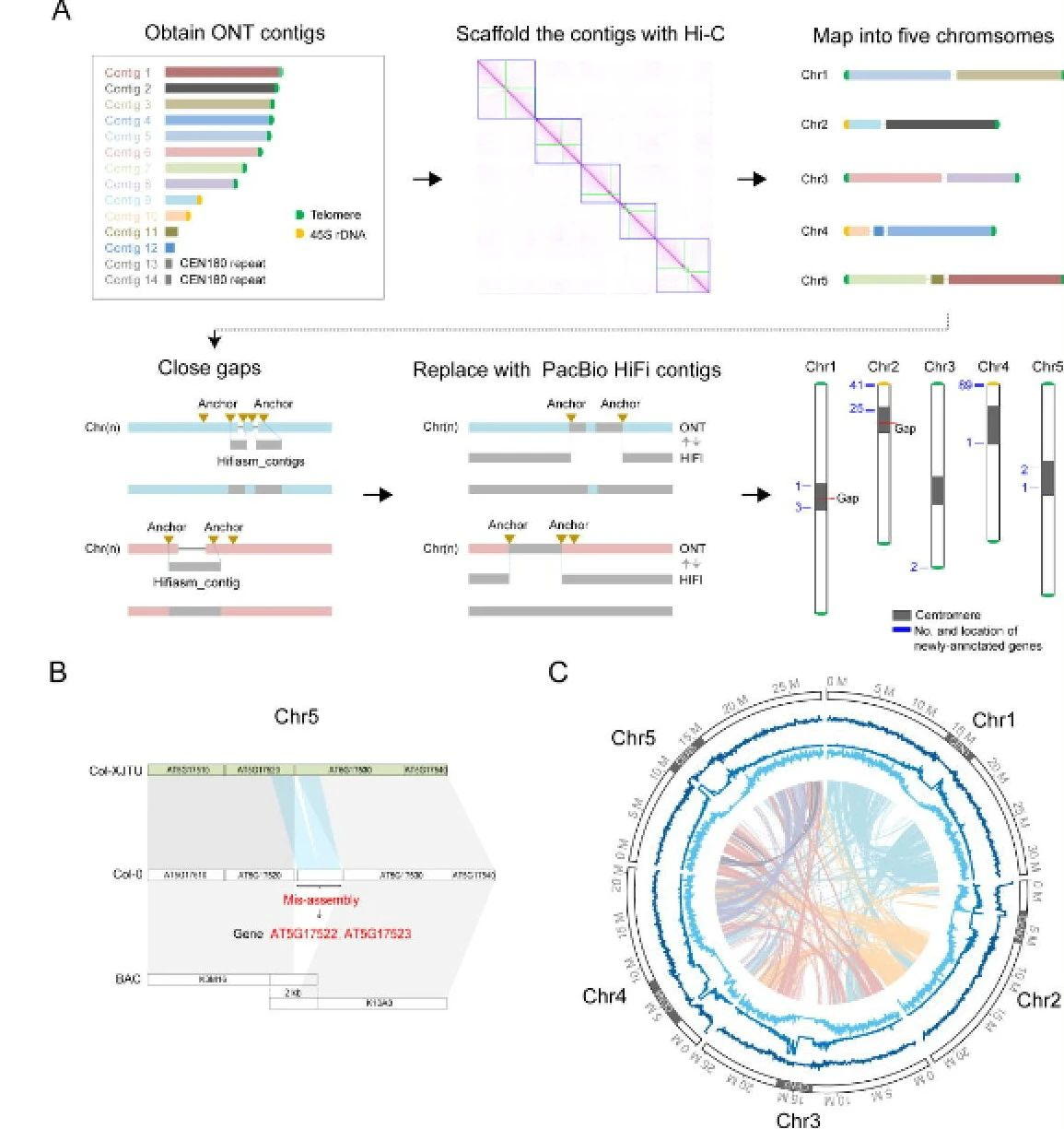
图4 拟南芥T2T组装策略
03 香蕉基因组近完成图

(一)
文章题目:Telomere-to-telomere gapless chromosomes of banana using nanopore sequencing
发表期刊:Communications Biology
完成图构建策略:ONT Ultra-Long+Bionano
研究结果:基于ONT 超长测序,研究者利用177×(其中17×>75Kb) 的数据构建了香蕉近完成图,其中有5条染色体为T2T组装。基于香蕉近完成图,研究者新发现了1700个基因,大部分位于1号和7号染色体上,这些新基因主要是串联重复基因,多以基因簇的形式存在于染色体重组区域,部分基因对香蕉的环境适应性和抗病性具有重要作用。此外,研究者还揭示了新组装的香蕉V4基因组与v1和v2版本间的共线性和结构变异,并对香蕉的A、B、S三套亚基因组的组装优化进行了初步探讨。总之,本研究构建了香蕉基因组的近完成图,并解析了着丝粒和同源基因簇等复杂区域的组成。
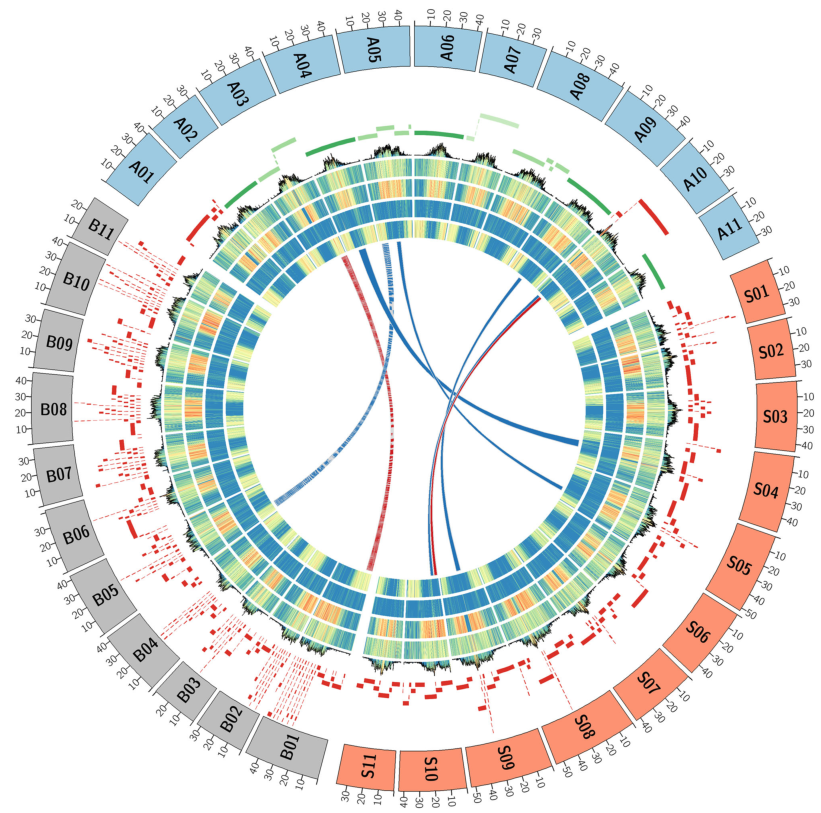
图5 香蕉基因组近完成图
04 澳洲胡桃基因组近完成图

(一)
文章题目:De novochromosome level assembly of a plant genome from long read sequence data
发表期刊:the plant journal
完成图构建策略:PacBio HiFi+Hi-C
研究结果:近年来,在植物基因组测序和组装方面的进展使得基因组的产生具有更高的连续性和序列准确性。本研究中,研究者通过PacBio HiFi测序并利用Hifiasm组装,在没有结合其它技术的情况下,就完成了澳洲胡桃近完成图的组装。澳洲胡桃的14条染色体中,有8条染色体仅由1个contig 组成(其中6条属于T2T),其余6条染色体由2-4个contig组成,这些断裂的区域主要是高度重复序列和核糖体基因,随后结合Hi-C技术构建染色体水平胡桃基因组,并对基因组中的重复序列和核糖体基因进行了研究。

图6 澳洲胡桃6条T2T染色体Contig版本与Hi-C版本间的共线性
05 大麦基因组近完成图

大麦是全球主要的粮食作物之一,关于其基因组测序的研究已有多篇报道。在2012年构建了大麦基因组草图后,2020年Nature 在线报道了20个大麦的泛基因组,但由于高重复高杂合等特点,大麦基因组完成图一直未有报道。
2021年3年,Plant Cell报道了“Long-read sequence assembly: a technical evaluation in barley”的研究论文,该研究通过对不同测序技术、组装软件进行比对,指出了PacBio HiFi技术在构建大麦基因组中连续性最好、准确度最高,随后结合Hi-C、Bionano、遗传图谱等技术构建了目前最完整的大麦基因组MorexV3(但仍然有1.32 Mb的Gap)。
2021年11月,bioRxiv报道了“Prospects of telomere-to-telomere assembly in barley: analysis of sequence gaps in the MorexV3 reference genome”的研究论文,该研究通过多种技术鉴定了大麦基因组中着丝粒、核糖体DNA和端粒重复序列的长度和频率分布,发现MorexV3几乎没有着丝粒序列和45S核糖体DNA重复序列,最后研究者指出ONT 超长技术可以为攻克这些gap提供较多帮助。
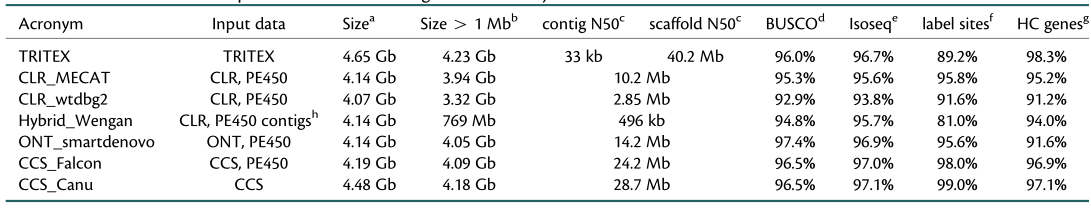

图7 大麦构建T2T基因组前的评估(上表测序技术,下图重复序列、着丝粒、端粒定位)
基因组完成图-动物篇
01 哺乳动物基因组完成图

随着测序与分析技术的突破,T2T联盟的科研人员先后组装发表了首个人类X染色体完成图、常染色体完成图、人类基因组完成图等,相关成果刊登在Nature期刊上。
在X染色体的完成图上,研究人员重建了近3.1 Mb的着丝粒卫星DNA阵列(DXZ1),并填补了GRCh38参考基因组上存在的29个缺口,其中包括来自人类假常染色体区域和癌症-睾丸两性基因家族(CT-X和GAGE)的新序列。
在8号染色体的完成图上,研究人员填补了5个空白区域,包含2.08 Mb的着丝粒α卫星阵列、β-防御素基因簇、染色体8q21.2位点上863 kb的可变数目串联重复序列(可作为新的着丝粒区域)。
在最终的首个人类基因组完成图中,研究者新增加或修正了238Mb的序列(该序列的大部分是由着丝粒卫星序列(180Mb)、重复片段(68Mb)和rDNAs(10Mb)组成),其中182Mb是全新的序列,并注释到2226个新基因。
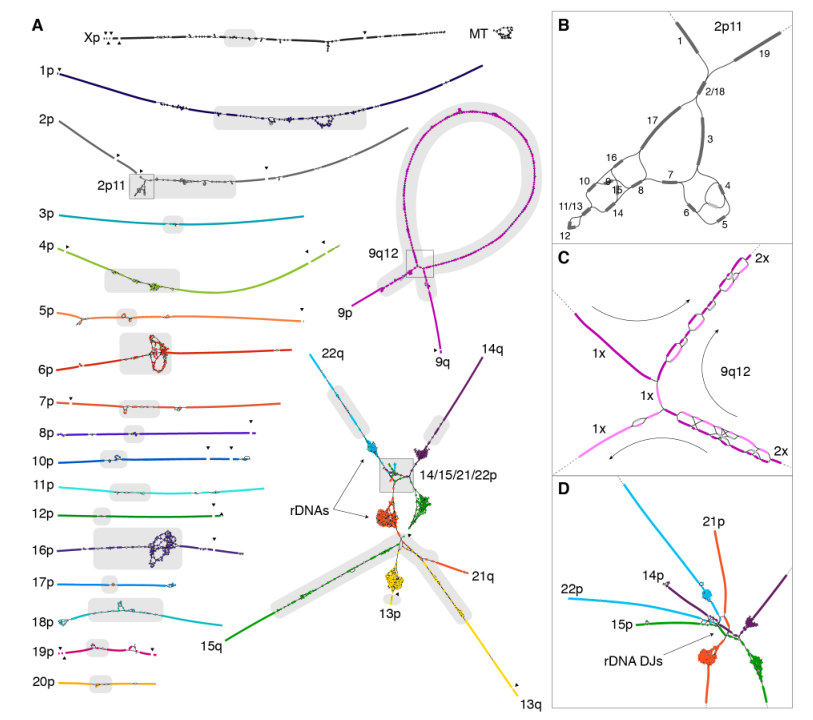
图8 HiFi组装人类基因组完成图步骤
02 鱼类基因组完成图

文章题目:Telomere-to-telomere assembly of a fish Y chromosome reveals the origin of a young sex chromosome pair
发表期刊:Genome Biology
完成图构建策略:PacBio HiFi+Hi-C
研究结果:在动物的染色体中,Y染色体以其基因含量少、高重复、高杂合等特征成为了最难以组装的区域之一。 本研究中,研究者通过HiFi和Hi-C技术构建了近乎完整的大刺鳅基因组,包括无间隙的Y染色体。随后基于重测序研究,研究者定位到了大刺鳅鱼Y染色体的性别决定区域SLR(7Mb),并通过比较X和Y染色体的SLR区域,证实了大刺鳅性染色体属于低重组类型。最后,研究者证实了HMGN6基因为大刺鳅的性别决定关键基因。
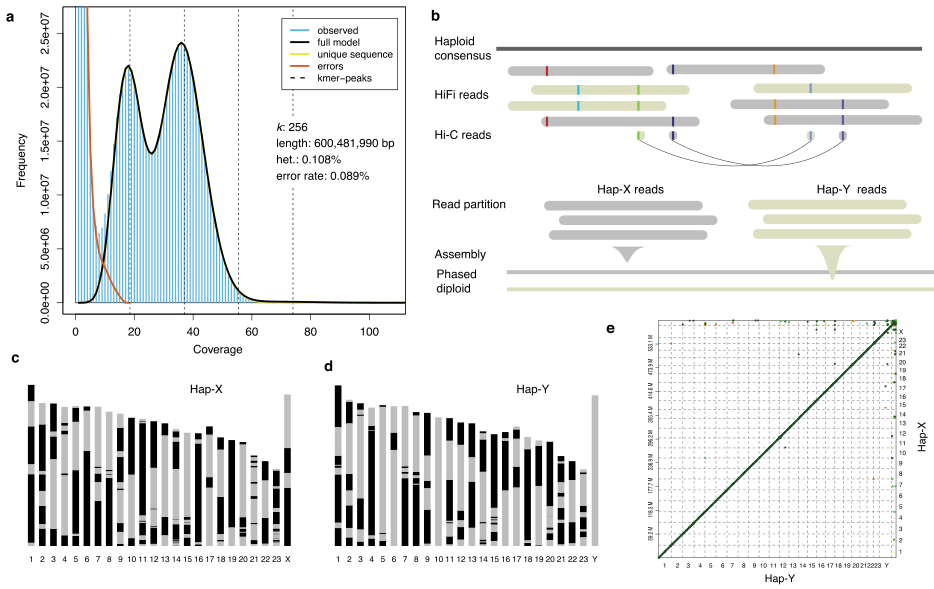
图9 大刺鳅基因组完成图的构建策略
03 虫类基因组完成图

文章题目:Telomere-to-telomere assembly of the genome of an individual Oikopleura dioica from Okinawa using Nanopore-based sequencing
发表期刊:BMC Genomics
完成图构建策略:ONT+Hi-C
研究结果:许多虫类基因组较小(小于100Mb),但GC含量极端且具有高重复特征。本研究中,研究者通过ONT和Hi-C技术构建了异体住囊虫的基因组完成图,获得的8条染色体中,有两条属于T2T,另外几条都至少包括了一个端粒,最终构建的基因组大小为64.3Mb,Contig N50=4.7Mb。此外,通过多种方法评估研究者证实了组装结果的可靠性,并基于新版本基因组更新了异体住囊虫的基因注释(发现了更多的基因)。总之,本研究构建的异体住囊虫基因组完成图可为其跨物种比较及适应性研究提供新见解。
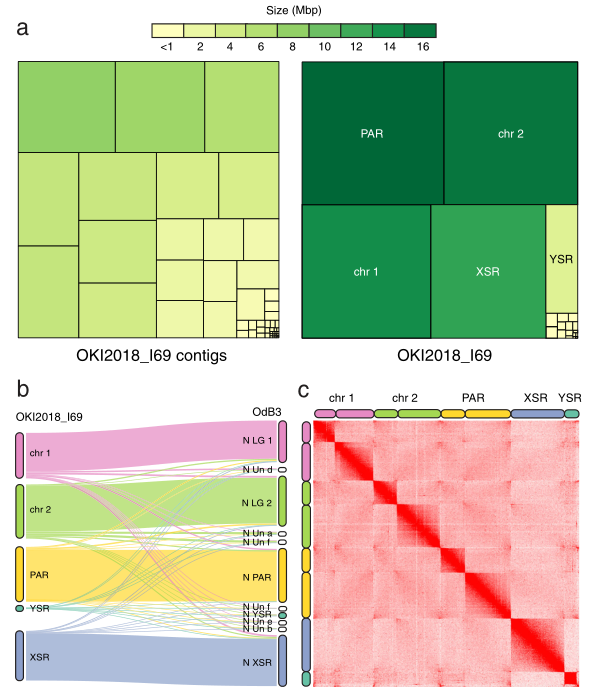
图10 异体住囊虫基因组完成图
总 结
通过对上述基因组完成图文章进行汇总和分析,我们不难发现,PacBio HiFi和ONT超长读长已经成为构建T2T基因组必不可少的基础测序技术,而Hi-C、Bionano、遗传图谱技术则可以将最终的基因组推向完成图水平。此外,在构建T2T基因组中,植物需要着重解决着丝粒、端粒和核糖体DNA序列,动物需要着重解决性染色体区域。
而在构建T2T基因组后,我们至少可以进行以下三方面的研究:
单个物种研究着丝粒结构和功能揭示着丝粒区域的染色体特征(需要结合转录组和表观多组学);多物种(近缘物种)着丝粒区域比较揭示其结构和功能的进化;
解析性染色体的起源、驯化以及鉴定性别分化关键基因;
全基因组比较揭示结构变异、新基因、重复序列等对物种进化的影响。
参考文献:
[1] Song J M, Xie W Z, Wang S, et al. Two gap-free reference genomes and a global view of the centromere architecture in rice[J]. Molecular plant, 2021, 14(10): 1757-1767.
[2] Li K, Jiang W, Hui Y, et al. Gapless indica rice genome reveals synergistic contributions of active transposable elements and segmental duplications to rice genome evolution[J]. Molecular Plant, 2021, 14(10): 1745-1756.
[3] Naish M, Alonge M, Wlodzimierz P, et al. The genetic and epigenetic landscape of the Arabidopsis centromeres[J]. Science, 2021, 374(6569): eabi7489.
[4] Wang B, Yang X, Jia Y, et al. High-quality Arabidopsis thaliana genome assembly with Nanopore and HiFi long reads[J]. Genomics, proteomics & bioinformatics, 2021.
[5] Belser, C., Baurens, FC., Noel, B. et al. Telomere-to-telomere gapless chromosomes of banana using nanopore sequencing. Commun Biol 4, 1047 (2021).
[6] Sharma P, Kharabian Masouleh A, Topp B, et al. Denovo chromosome level assembly of a plant genome from long read sequence data[J]. The Plant Journal, 2021.
[7] Mascher M, Wicker T, Jenkins J, et al. Long-read sequence assembly: a technical evaluation in barley[J]. Plant Cell, 2021: Epub ahead of print.
[8] Nurk S, Koren S, Rhie A, et al. The complete sequence of a human genome[J]. bioRxiv, 2021.
[9] Xue L, Gao Y, Wu M, et al. Telomere-to-telomere assembly of a fish Y chromosome reveals the origin of a young sex chromosome pair[J]. Genome biology, 2021, 22(1): 1-20.
[10] Bliznina A, Masunaga A, Mansfield M J, et al. Telomere-to-telomere assembly of the genome of an individual Oikopleura dioica from Okinawa using Nanopore-based sequencing[J]. BMC genomics, 2021, 22(1): 1-18.
 微信公众号
微信公众号
