
研究基因组进化的过程和机制,对于从基因组水平理解遗传学和物种多样性至关重要。酿酒酵母(Saccharomyces cerevisiae)是研究真核基因组进化的强大模式生物,已经被用于比较基因组学的研究,揭示了一些机制。这些研究依赖于相对静态的基因组序列,可能遗漏了许多过程动态的细节。合成基因组研究极大地促进了基因组进化和工程的研究,例如基因组最小化、遗传密码重编码、引入合成组分和数据存储。在最近被用于研究基因组进化的方法中,SCRaMbLE系统可以在几乎所有非必需基因的3′ UTR中放置对称的loxP位点(loxPsym)。而且Cre重组酶活性可以快速触发loxPsym位点之间的重组,产生结构变异,包括缺失、倒位、重复和易位。
近期,天津大学元英进教授研究团队在国际知名期刊National Science Review(IF:23.178)以“Dynamics of synthetic yeast chromosome evolution shaped by hierarchical chromatin organization ”为题发表合成基因组方向研究成果。该研究通过人工基因组重排技术,创制了超过26万种人工染色体重排的酵母种质资源库,解析基因组重排规律,为高效定向培养微生物菌株奠定了基础。该研究将5.5个合成染色体(synII、synIII、synV、环形synVI、synIXR和synX)整合在一个单倍体菌株中,并建立了一个位点特异性重组系统,使得能够消除相应的天然染色体。在这个具有近四分之一合成基因组的菌株中,该研究通过SCRaMbLE(Synthetic Chromosome Rearrangement and Modification by LoxP-mediated Evolution)系统,生成了大量涉及染色体内和染色体间重组的重排事件。该研究对经过SCRaMbLEd的细胞进行基因组测序,并通过分析loxPsym位点检测到260000个以上重排事件。通过多种数据分析每个重排事件,该研究揭示了合成染色体之间稳定的重排图谱,并且这种重排与局部染色质状态(ATAC-seq)和三维基因组结构(Hi-C)相关。该研究为层级染色质组织对基因组进化动态的组合效应提供了见解。

研究思路

研究结果
1.合成型酵母染色体的特定重排模式
该研究通过多个步骤构建了多条合成染色体的酵母菌株。首先,将两个不同配型的单倍体菌株(yYW169和yZY192)杂交,得到一个含有6条合成染色体和6条天然染色体的杂合子菌株(yYW268),并基于Vika/vox重组系统(一种与Cre/loxP正交的位点特异性重组系统 )得到一个只含有6条合成染色体的二倍体菌株(yYW393)。通过孢子化和四分体分离,研究获得含有6条合成染色体的单倍体菌株(yYW394)。yYW394的基因组约为2.61 Mb(酵母基因组大小的~22.0%)。随后通过适应性实验室进化,得到了一个进化后的菌株(yZSJ025)。
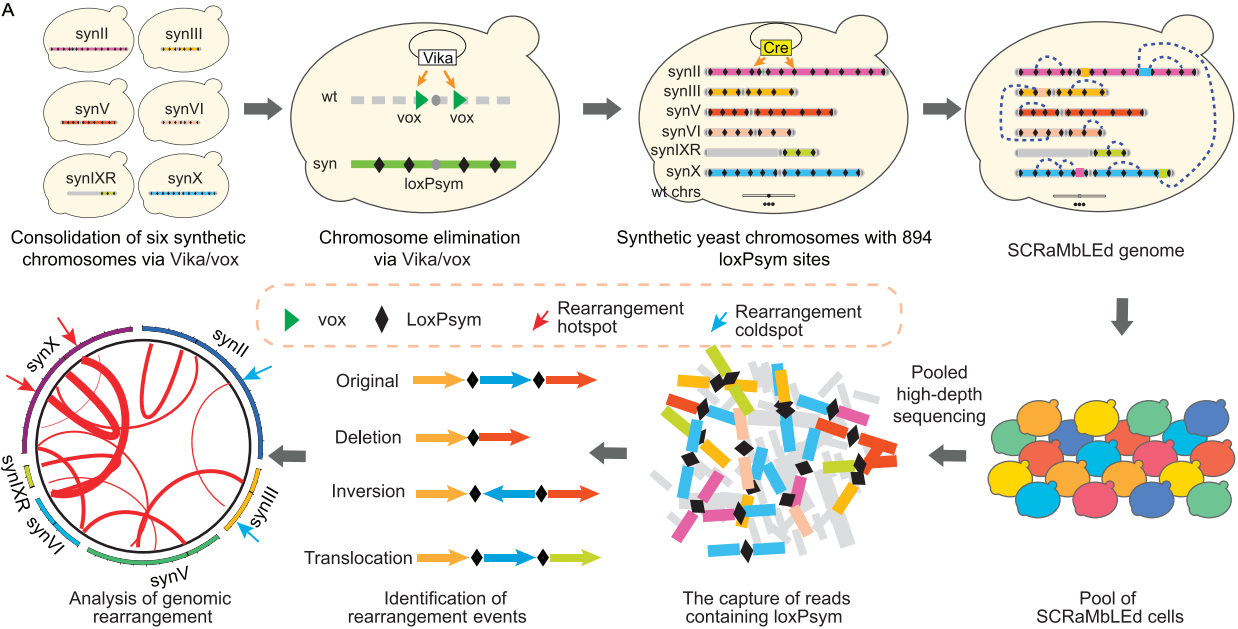
图1.合成酵母染色体的步骤以及SCRaMbLE引发染色体重排
接下来实施SCRaMbLE,将表达Cre重组酶的质粒转化到含有894个loxPsym位点的进化菌株yZSJ025中,并加入β-雌二醇激活Cre重组酶的活性,使菌株合成染色体发生重排。随后,研究对SCRaMbLE后的菌株进行深度测序,并通过筛选和比对含有loxPsym序列的reads识别和分类重排事件。合成染色体中共有263520个重排事件,其中包括124499个染色体内部的重排事件和139021个染色体间的重排事件。前者含有62106个倒位、22526个缺失和39867个复杂的重排事件。同时,与染色体内重排相比,染色体间重排的频率相对较低。并且每个染色体的重排数量与loxPsym位点数量正相关,每条染色体上的loxPsym位点有相似的重排概率。
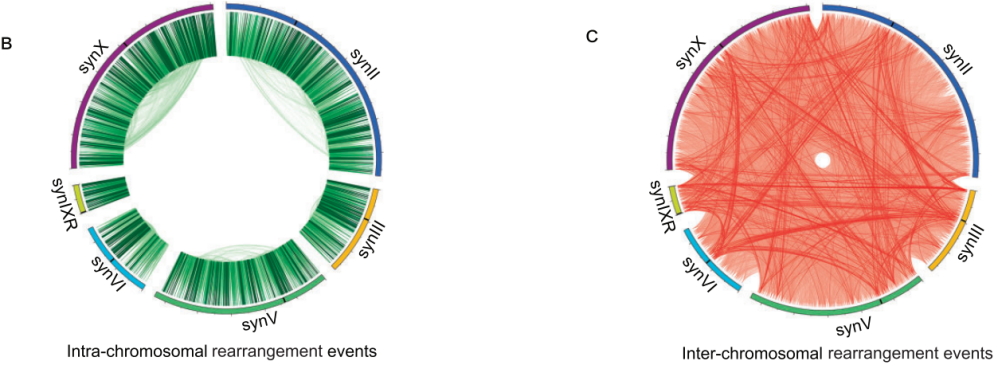
图2.合成染色体中染色体内和染色体之间的重排
该研究根据每个loxPsym位点附近的重排reads来计算每个位点的重排频率,并将所有877个位点按照重排频率分成了十个等级。该研究分别定义了最高等级和最低等级的88个位点为重排热点(hotspot)和重排冷点(coldspot)。研究发现在不同的SCRaMbLE实验中,合成染色体上的重排频率分布是高度一致的,说明染色体上有一些区域天生就更容易或更难发生重排。研究比较了不同酵母菌株中相同合成染色体的重排模式,发现合成染色体内部在不同菌株中都表现出相似的染色体内部重排模式。最后结合RNA-seq分析,研究发现基因转录水平和相邻重组频率之间没有明显的相关性。
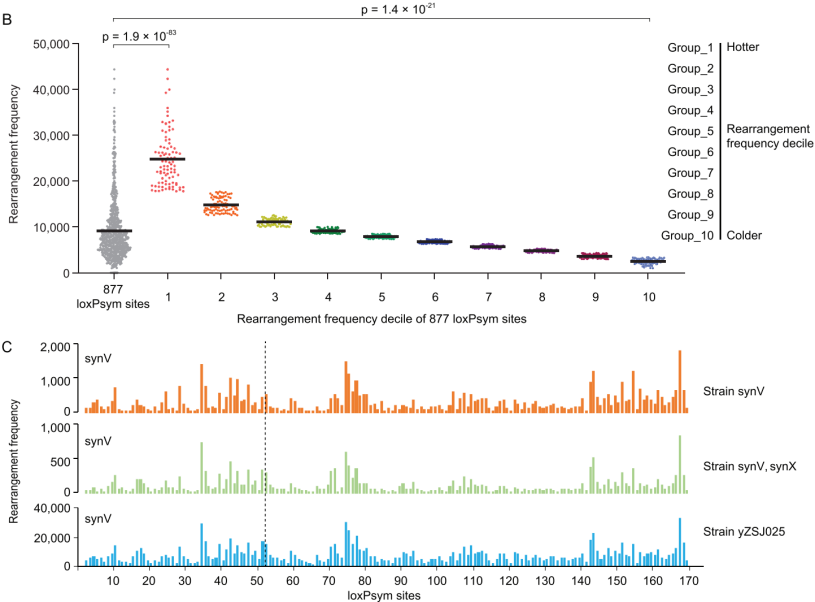
图3.重排频率分级(上)和不同酵母菌株中相同合成染色体的重排模式(下)
2.重排频率与染色质开放性相关
SCRaMbLE方法需要Cre重组酶和loxPsym位点之间存在相互作用。该研究推测重排频率可能受到染色质开放性的影响,并利用ATAC-seq方法测量了yZSJ025菌株的全基因组染色质开放性。该研究分析了每个loxPsym位点附近的ATAC-seq信号,发现ATAC-seq信号强度与重排频率有很好的相关性,即重排频率越高的位点,染色质开放性越高,反之亦然。例如,在合成染色体synX,重排冷点SET4 3' UTR区域的loxPsym 位点的ATAC-seq信号很弱,重排热点PRY3 3' UTR区域的loxPsym位点的ATAC-seq信号很强。该研究还利用核小体占据率分析证实了这一结果,即重排热点位点的核小体占据率较低,而重排冷点位点的核小体占据率较高。最后,该研究比较合成酵母和天然酵母在相应区域的染色质开放性,发现它们是一致的,说明合成酵母基因组中引入的loxPsym位点和其他序列改造没有影响染色质开放性。这也说明了合成酵母基因组在表观遗传水平上与天然酵母基因组是相似的。
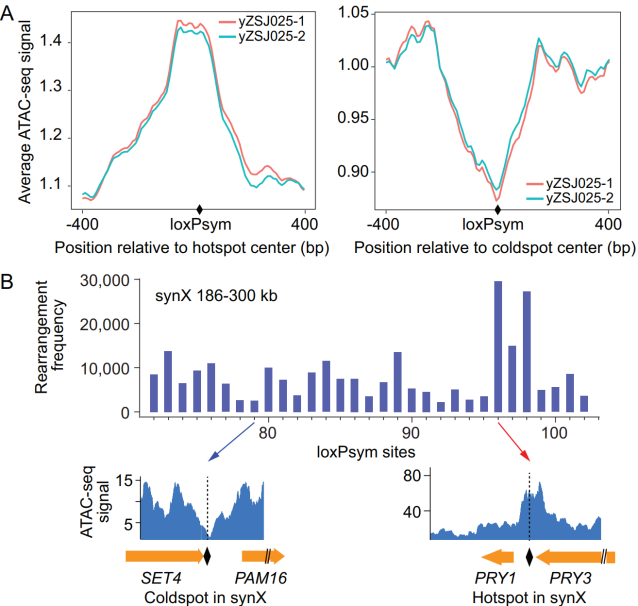
图4.重排频率和染色质开放性相关(上)及在合成染色体synX的相应示例(下)
3.重排频率和染色质构象有关
接下来,研究利用Hi-C测量了yZSJ025菌株的全基因组三维结构,得到了合成染色体不同基因组区域之间的互作频率,并绘制重排频率热图以将其与互作频率进行对比。研究发现,重排频率与互作频率有很好的一致性。在热图中,对角线附近的区域代表染色体上相邻的位点。这些染色体内部的区域在互作热图和重排频率热图中表现有最高的互作频率和最高的重排频率。而且重排事件更倾向于发生在合成染色体上相邻的loxPsym位点之间。此外,染色体合成酵母中环状染色体提供了机会探究环状染色体和线性染色体在细胞中发生重排的差异。研究发现,与其他线性染色体(synV)相比,环状染色体(synVI)上两个端粒区域之间的重排频率更高。

图5.重排频率与互作频率的一致性(上)及环状染色体上两个端粒区域之间的重排频率更高(下)
研究者还注意到染色体上的着丝粒区域和端粒区域在不同染色体之间有较高的互作频率,这与它们在细胞核中聚集在一起的特点是一致的。相应地,着丝粒区域和端粒区域在不同染色体之间也有较高的重排频率。综上,重排事件通常更容易发生在空间距离更近的基因组区域之间。

图6.合成染色体(彩色)和天然染色体(灰色)的3D结构模型
4.染色质开放性和染色体构象的协同效应
根据前面的结果,研究者提出了一个假设,即染色质开放性和空间距离对重排频率有协同的影响,而不是独立的影响。研究者根据每对loxPsym位点之间的重排事件来检验他们的假设,发现如果两个位点的染色质开放性相近,那么重排频率主要受到它们的空间距离的制约;如果两个位点的空间距离相近,那么重排频率主要受到它们的染色质开放性的影响。 此外,如果两个位点的染色质呈开放状态,但是空间距离很远,那么它们的重排频率与另一些染色质闭合但空间距离很近的位点的重排频率相似。也就是说,染色质开放性对重排频率的影响可以被空间距离的差异所抵消,反之亦然。总之,该研究认为酵母基因组中的染色质结构和空间构象在SCRaMbLE过程中以一种协同的方式影响着重排频率。

图7.染色质开放性和染色体构象的协同效应
小 结
该研究在一个含有5.5个合成酵母染色体的酵母菌株检测到260000个以上重排事件。值得注意的是,重排事件呈现出一种特定的频率。该研究通过进一步分析揭示了这种重排频率景观是由染色质开放性和空间互作频率的组合效应塑造的。重排倾向于发生在三维空间上接近和染色质开放的区域。SCRaMbLE介导的大量重排事件为引导基因组进化提供了动力,而对重排景观的研究为基因组进化动态提供了深入的机制见解。该研究展示了人工基因组重排技术作为定向基因组进化工具的独特能力,加速了遗传多样性产生过程,为培育高性能微生物菌株提供了技术基础。此外,酵母基因组重排规律的解析为研究染色质结构对染色体重排的影响提供了机制性的见解,为系统地探索真核生物基因组动态进化提供了一种新的视角,并且为更高版本的基因组设计合成提供了理论参考。
天津大学元英进教授为本文的通讯作者,天津大学周嗣杰和吴毅为本文共同第一作者,永利集团3044刘琳博士署名参与了本研究。本研究得到了国家重点研发计划 (2021YFC2100800)和国家自然科学基金 (21621004、31861143017和31971351)的支持。
参考文献:
Zhou S, Wu Y, Zhao Y, et al. Dynamics of synthetic yeast chromosome evolution shaped by hierarchical chromatin organization[J]. National Science Review, 2023, 10(5): nwad073.
 微信公众号
微信公众号
